¿Qué son los polimorfismos de un solo nucleótido?

Las dos formas más frecuentes de variación del ADN del genoma humano son los polimorfismos de un solo nucleótido (SNP) y las variaciones en el número de copias (CNV).
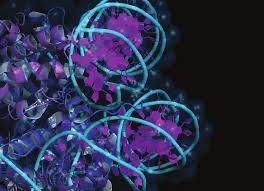
Los polimorfismos de un solo nucleótido (SNP, por sus siglas en inglés) son variaciones genéticas comunes que se encuentran en el ADN de los individuos. Un SNP es un cambio en una sola base de ADN en una determinada posición del genoma. Estos cambios pueden involucrar la sustitución de una base por otra (por ejemplo, A por T) o la inserción o eliminación de una base.
Los SNP son la forma más común de variación genética en los seres humanos y se encuentran dispersos por todo el genoma. Pueden ocurrir en regiones codificantes de genes, donde pueden afectar la secuencia de aminoácidos y, por lo tanto, la estructura y función de las proteínas resultantes. También pueden estar presentes en regiones no codificantes del ADN, como los intrones y las regiones intergénicas, donde pueden influir en la regulación de la expresión génica
Los SNP son variantes de un solo nucleótido y casi siempre son bialélicos (solo existen dos opciones para un lugar determinado en la población, como A o T). Se han identificado más de 6 millones de SNP humanos y muchos de ellos tienen una amplia variabilidad en la frecuencia en las distintas poblaciones. Se deben destacar las siguientes
características:
- Existen SNP en todo el genoma, dentro de los exones, intrones, regiones intergénicas y regiones codificantes.
- Apenas un 1 % de los SNP se localizan en regiones codificantes, que es una cifra que cabría esperar por azar dado que las regiones codificantes representan
aproximadamente un 1,5% del genoma. - Los SNP localizados en regiones no codifican tes pueden afectar a elementos reguladores del genoma, de forma que modifican la expresión de los genes; en este caso los SNP pueden influir directamente sobre la susceptibilidad a la enfermedad.
- Pueden existir variantes «neutras» de SNP sin efectos sobre la función de los genes o el fenotipo portador.
- Incluso los SNP «neutros» pueden ser marcadores útiles si se coheredan con un gen asociado a enfermedad como consecuencia de la proximidad física. Dicho de otromodo, los SNP y el factor genético causante se encuentran en desequilibrio de ligamiento.
- El efecto de la mayor parte de los SNP sobre la susceptibilidad a la enfermedad es débil, pero todavía se tiene que valorar si se puede emplear la identificación de estas variantes, solas o combinadas, para desarrollar estrategias eficaces para la predicción o prevención de enfermedades.
 Síguenos en X: @el_homomedicus y @enarm_intensivo
APRENDER CIRUGÍA
Síguenos en X: @el_homomedicus y @enarm_intensivo
APRENDER CIRUGÍA