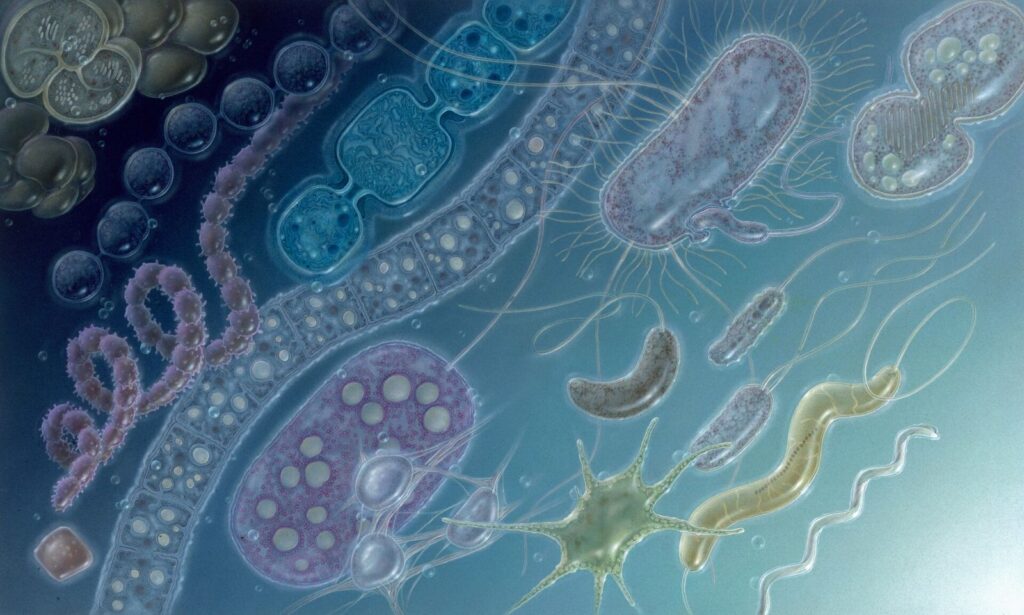
La clasificación de las bacterias constituye un pilar fundamental en microbiología, ya que permite diferenciar organismos que, aunque microscópicamente invisibles a simple vista, poseen una diversidad estructural, funcional y genética extraordinaria. La taxonomía bacteriana se realiza utilizando criterios variados que incluyen características macroscópicas y microscópicas, propiedades de crecimiento y metabólicas, antigénicas y, más recientemente, genéticas. Cada enfoque proporciona un ángulo distinto de observación y, al combinarse, permite a los investigadores y clínicos identificar bacterias de manera precisa y establecer estrategias diagnósticas y terapéuticas adecuadas.
Distinciones macroscópicas y microscópicas
Características macroscópicas
El análisis macroscópico de las bacterias se basa principalmente en la observación de sus colonias cuando se cultivan en medios nutritivos y selectivos. Una colonia representa una comunidad microbiana compuesta por millones de células derivadas de un solo organismo progenitor. Este conglomerado ofrece rasgos visibles que facilitan la identificación inicial: forma (circular, irregular, filamentosa), tamaño (milimétrico o submilimétrico), textura (lisa, rugosa, viscosa), coloración (blanca, amarilla, roja, verde) y, en algunos casos, emisión de olores característicos debido a metabolitos volátiles producidos por la bacteria.
Además, las bacterias pueden demostrar propiedades metabólicas específicas cuando se exponen a diferentes sustratos o condiciones de crecimiento. Por ejemplo, la capacidad de fermentar determinados azúcares, como la lactosa, permite diferenciar especies intestinales como Escherichia coli, que fermenta lactosa, de Salmonella, que no lo hace. La actividad hemolítica, observada en medios con sangre, distingue especies de Streptococcus según el tipo de lisis de los eritrocitos (alfa, beta o gamma hemólisis). Asimismo, la capacidad de secretar enzimas como lipasas o proteasas, presentes en géneros como Clostridium o Pseudomonas, evidencia diferencias funcionales que pueden ser críticas para el diagnóstico clínico.
Características microscópicas
La observación microscópica permite analizar la morfología celular, la disposición de las bacterias y su reacción frente a colorantes específicos. La forma de la célula bacteriana —coco, bacilo, espirilo o curva— constituye un criterio inicial de identificación. La organización de estas células en cadenas, racimos, diplococos o estructuras ramificadas añade información adicional sobre la especie. Por ejemplo, Staphylococcus aureus se organiza típicamente en racimos semejantes a uvas, mientras que Streptococcus pyogenesforma largas cadenas lineales. Las especies filamentosas como Nocardia o Actinomycesimitan la apariencia de los hongos, lo que refleja una complejidad estructural única dentro del mundo bacteriano.
Uno de los métodos más utilizados para diferenciar bacterias a nivel microscópico es la tinción de Gram, que permite clasificar los organismos en Grampositivos o Gramnegativos en función de la estructura de su pared celular. Este procedimiento, rápido y altamente informativo, implica la fijación de las células, la aplicación de cristal violeta y yodo, la decoloración con acetona-alcohol y el contraste con safranina. Las bacterias Grampositivas retienen el colorante violeta debido a su gruesa capa de peptidoglicano, adoptando un color púrpura intenso. Por el contrario, las Gramnegativas, con una pared más delgada, pierden el cristal violeta durante la decoloración y se tiñen de rojo por la safranina. Este método no solo facilita la identificación inicial, sino que también orienta la elección de antibióticos, dado que la estructura de la pared celular influye en la susceptibilidad a fármacos específicos.
Es importante destacar que la tinción de Gram puede fallar en condiciones especiales. Bacterias en cultivos envejecidos o en estado de estrés metabólico pueden perder la capacidad de retener el colorante. Algunas especies, como las micobacterias, poseen paredes ricas en lípidos que requieren tinciones alternativas como la ácido-alcohol resistente, mientras que los micoplasmas carecen de peptidoglicano y no se tiñen con los métodos tradicionales. Estas excepciones subrayan la necesidad de combinar múltiples criterios en la clasificación bacteriana para lograr una identificación precisa.
Distinción metabólica, antigénica y genética
La clasificación bacteriana no se limita a la observación de la morfología celular y de las colonias; niveles más avanzados se basan en la firma metabólica, antigénica y genética de cada organismo. Estas características reflejan no solo la estructura interna y externa de la célula, sino también sus adaptaciones funcionales a diferentes ambientes y su capacidad para producir compuestos específicos que son útiles tanto para la identificación como para la comprensión de su fisiología y patogenicidad.
Clasificación metabólica
El perfil metabólico de una bacteria constituye un conjunto de rasgos funcionales que permiten diferenciar especies y cepas. Entre estos rasgos se incluyen la capacidad de tolerar o resistir ciertos compuestos químicos, como detergentes y ácidos biliares, que afectan la integridad de la membrana celular; la exigencia de condiciones específicas de oxígeno, distinguiendo bacterias aeróbicas, anaeróbicas o microaerófilas; y la necesidad de nutrientes particulares, como aminoácidos, vitaminas o carbohidratos específicos, para sostener el crecimiento.
Adicionalmente, las bacterias pueden producir metabolitos característicos, incluyendo ácidos orgánicos, alcoholes, gases o pigmentos, que reflejan rutas bioquímicas particulares. La actividad enzimática también constituye un marcador útil: por ejemplo, la producción de catalasa en estafilococos permite distinguirlos de los estreptococos, mientras que otras enzimas, como la ureasa o la citrato permeasa, facilitan la identificación de enterobacterias. Para el análisis sistemático de estas propiedades, se han desarrollado procedimientos automatizados que evalúan el crecimiento en distintos sustratos y condiciones, generando un biotipo numérico que representa el patrón metabólico de cada aislado bacteriano.
Clasificación antigénica
La serotipificación o clasificación basada en antígenos es un método que permite identificar bacterias mediante el reconocimiento de moléculas específicas presentes en su superficie, como polisacáridos de la pared celular, proteínas de membrana o flagelos. Esta estrategia es particularmente útil para bacterias que son difíciles de cultivar en laboratorio, peligrosas de manipular o asociadas con síndromes clínicos concretos. Por ejemplo, Treponema pallidum, el agente causal de la sífilis, y Francisella tularensis, responsable de la tularemia, se detectan con anticuerpos específicos debido a las dificultades de crecimiento o riesgo que presentan.
La serotipificación también se emplea para subdividir bacterias por debajo del nivel de especie, lo que tiene relevancia epidemiológica. Un ejemplo relevante es Escherichia coliO157:H7, un serotipo productor de toxinas que causa colitis hemorrágica, cuya rápida identificación es esencial para prevenir brotes. Del mismo modo, la identificación de Streptococcus pyogenes mediante antígenos específicos permite iniciar un tratamiento temprano de la faringitis estreptocócica y evitar complicaciones.
Clasificación genética
El análisis genético representa el método más preciso y definitivo para clasificar bacterias, ya que examina directamente la información codificada en el ADN o las proteínas del organismo. Las técnicas modernas incluyen la hibridación de ADN, la amplificación mediante reacción en cadena de la polimerasa y la secuenciación de ácidos nucleicos, así como la espectrometría de masas para determinar perfiles proteicos característicos (como en la técnica MALDI-TOF).
Estas estrategias permiten identificar bacterias sin necesidad de cultivarlas, lo que resulta fundamental para organismos de crecimiento lento, como las micobacterias, o altamente virulentos, que representan riesgos para el personal de laboratorio. La secuenciación de regiones conservadas del ADN ribosomal permite establecer relaciones evolutivas a nivel de familia, género, especie o subespecie, mientras que el análisis de genes específicos proporciona información sobre cepas individuales. Gracias a los avances tecnológicos recientes, estas técnicas se han simplificado, estandarizado y abaratan, de modo que se han incorporado rutinariamente en la práctica clínica y en estudios epidemiológicos a nivel global.

Fuente y lecturas recomendadas:
- Madigan, M. T., Martinko, J. M., Bender, K. S., Buckley, D. H., & Stahl, D. A. (2018). Brock biology of microorganisms (15th ed.). Pearson.
- Murray, P. R., Rosenthal, K. S., & Pfaller, M. A. (2025). Medical microbiology (10th ed.). Elsevier.
- Carroll, K. C., & Pfaller, M. A. (2023). Manual of clinical microbiology (13th ed.). American Society for Microbiology Press.
- Riedel, S., Hobden, J. A., Miller, S., Morse, S. A., Mietzner, T. A., Detrick, B., Mitchell, T. G., Sakanari, J. A., Hotez, P., & Mejía, R. (2020). Microbiología médica (28ª ed.). McGraw-Hill Interamericana Editores.